2 湖南农业大学生物信息系, 长沙, 410128
 作者
作者  通讯作者
通讯作者
计算分子生物学, 2012 年, 第 1 卷, 第 6 篇
收稿日期: 2012年10月27日 接受日期: 2012年10月28日 发表日期: 2012年10月29日
Cui Y., Zhu J.Z., Zhou Q., Jiang J., and Gao B.D., 2012, Sequence analysis of genome segment S10 of Hunan Dingcheng isolate of Southern Rice Black-Streaked dwarf virus, Jiyin Zuxue Yu Yingyong Shengwuxue (Genomics and Applied Biology), 31(2): 160-166 (崔亚, 朱俊子, 周倩, 蒋坚, 高必达, 2012, 水稻南方黑条矮缩病毒湖南鼎城株系S10片段的基因组序列分析, 基因组学与应用生物学, 31(2): 160-166)
运用 RT-PCR 技术克隆了水稻南方黑条矮缩病毒(southern rice black-streaked dwarf virus, SRBSDV) 湖南鼎城株系的基因组 S10 片段(SRBSDV-HuNDC S10),并对其全序列进行了测定和生物信息学分析。结 果显示,SRBSDV-HuNDC S10 片段全长为 1 797 bp (登录号: JQ337964),含有 1 个 ORF,编码 557 个氨基 酸残基的衣壳蛋白,推测分子量约 62.6 kD,推测等电点为 7.62,与已报道的广东、海南和云南分离物病毒 的 S10 作比较,它们的核苷酸相似性分别为 99.7%、99.0%和 98.4%,氨基酸相似性分别为 100.0%、99.5% 和 99.3%。对 SRBSDV-HuNDC S10 及部分 Fijiviruses 病毒对应片段在 5' URT 与 3' URT 存在的保守序列 和互补序列进行了归纳,对其 ORF 编码的氨基酸序列进行了 motif 查找,得到该属(Fijiviruses)氨基酸序列 的 10 个保守区段。此外,进行了糖基化位点、磷酸化位点及 B 细胞抗原表位预测,发现了 3 个可能的 N 端 豆蔻酰基化位点,可能与病毒的侵染机制有关。
南方水稻黑条矮缩病的大面积流行给我国南部 和越南北部水稻生产造成极大的危害。自2001年在我国广东省阳西县晚稻上首次发现以来(周国辉等, 2010),扩散十分迅速,给我国南方各省中、晚稻生产造成严重损失。2009年,该病在广东、海南、江西和湖南等省的部分稻区爆发,据不完全统计,造成约6 500 hm2水稻失收,受害面积至少达3×105 hm2(周国辉等, 2010)。
南方水稻黑条矮缩病毒(southern rice black-stre-aked dwarf virus, SRBSDV)是呼肠孤病毒科(Reoviridae)斐济病毒属(Fijivirus)成员之一,为该属的一个建议新种(Zhou et al., 2008; Zhang et al., 2008)。与RB-SDV(rice black-streaked dwarf virus)、MRDV(maize rough dwarf virus)、MRCV(Mal de Rio Cuarto virus)、OSDV(oat sterile dwarf virus)、FDV(Fiji disease virus)、NLRV(Nilaparvata lugens reovirus)同属(Milne et al., 2005),具有较近的亲缘关系。SRBSDV基因组由10条dsRNA片段组成,按在聚丙烯酰胺凝胶电泳中的迁移率由大到小分别命名为S1到S10(Zhou et al., 2008)。最外层外壳蛋白由S10编码(Wang et al., 2010),其序列具有保守性,是分类和鉴定病毒属种的重要依据。
迄今为止,NCBI数据库中有5条S10全序列,广东(EU784840) (Wang et al., 2010)、海南(EU523360 和NC_014713) (Wang et al., 2010)、云南(HQ394212)和山东(GQ472845) (Yin et al., 2011),广东和海南更 是完成了全基因组测序(Wang et al., 2010)。然而,对S10序列的基因结构并没有详细的报道。S10编码的最外层衣壳蛋白是否与病毒致病性、病毒的寄主选择或致使寄主产生某些病症有关,尚不清楚。
本研究是在完成SRBSDV S10湖南分离物的测定的基础上,利用生物信息学方法对其进行序列结构特征和同源性分析。
1 结果与分析
1.1 SRBSDV检测
利用本实验室已获专利的SRBSDV检测引物(周倩等, 2010),对湖南省常德植保站送检水稻进行了检测,出现约500 bp条带,为阳性。
1.2 全序列分析
测定的SRBSDV中国湖南常德鼎城分离物S10片段全长为1 797 bp(GenBank 登录号为 JQ337964),GC含量为35.56%,其唯一的ORF编码的蛋白推测分子量为62.6 kD(表1)。与已报道的广东、海南和云南分离物病毒的S10全序列相比,它们的核苷酸相似性分别为99.7%、99.0%和98.4%,氨基酸相似性分别为100.0%、99.5%和99.3%(表2)。与海南分离物相比,在20处碱基突变中C/T间12次,A/G间的转变5次,A/T间1次,G/T间1次,1 788位空1次;编码区突变12次,3' URT突变8次,5' URT突变0次。同类碱基之间的转换(即C/T, A/G)共17次,占总突变的85%。
|
表1 SRBSDV-S10 的湖南, 广东, 海南, 云南株系和部分Fijiviruses 病毒对应片段比较
Table 1 Comparison of SRBSDV-S10 and SRBSDV of HuNDC, GD, HaN, YN isolates and other related Fijiviruses
|
|
表2 湖南, 广东, 海南, 云南SRBSDV S10 和部分Fijiviruses 病毒对应片段核苷酸和氨基酸序列的相似性比较
注: 左下角显示核酸相似性; 右上角显示氨基酸相似性
Table 2 Nucleotide and putative amino acid identity among SRBSDV S10 of HuNDC, GD, HaN and YN isolates and corresponding segments of the related Fijiviruses
Note: Down-left shows nucleotide identity; Upper-right shows putative amino acid identity
|
S10两端存在保守序列(方框内)和紧接保守序列的不完全反向互补序列(下划线)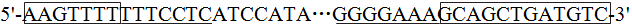 (Zhou et al., 2008; Zhang et al., 2008; Wang et al., 2010)。其它亲缘关系相近的病毒相应片段也有类似结构,但OSDV和NLRV除外(表1)。SRBSDV病毒S1~S9也存在相似结构(Wang et al., 2010)。这些结构可能在病毒的复制和翻译相关蛋白的调节过程中发挥功能。
(Zhou et al., 2008; Zhang et al., 2008; Wang et al., 2010)。其它亲缘关系相近的病毒相应片段也有类似结构,但OSDV和NLRV除外(表1)。SRBSDV病毒S1~S9也存在相似结构(Wang et al., 2010)。这些结构可能在病毒的复制和翻译相关蛋白的调节过程中发挥功能。
1.3 S10 编码蛋白的motif分析
S10仅编码一个长为557个氨基酸残基的ORF,推测分子量约62.6 kD,推测等电点为7.62,编码病毒的最外层衣壳蛋白。对该属相关病毒的10条外层衣壳蛋白氨基酸序列进行MEME软件在线分析,找到10个保守区域。10个保守区域在10条序列上均存在,而且覆盖序列的很大部分(图1),进一步证明该病毒属衣壳蛋白序列高度保守。就保守区域的位置而言,OSDV、FDV、NLRV与其它有所不同,而其它 7 条则基本一致。将10 个motif的匹配模式整理为表3。
|
图1 全部保守区域
Figure 1 Combined block diagrams
|
|
表3 10 个保守区域的匹配模式
Table 3 Regular expression of the ten motifs
|
1.4系统发育树分析
两个发育树都显示湖南病毒分离物与广东、海南、云南SRBSDV病毒分离物在同一分支上,进一步证明了湖南鼎城病毒分离物为SRBSDV的一个地理小种(图2)。
|
图2 SRBSDV S10 全序列系统发育树
Figure 2 Phylogenetic tree of complete segment of SRBSDV S10
|
1.5糖基化位点、磷酸化位点及B细胞抗原表位预测
湖南鼎城SRBSDV分离物S10编码的蛋白含有8个糖基化位点:87-NITQ、114-NLSD、152-NVTL、253-NISL、364-NTSG、370-NITK、454-NLTI和536-NSST,其中,第364位和第536位可能性相对较小。磷酸化位点:丝氨酸(Ser)有22个位点(37, 51, 97, 116, 128, 133, 159, 227, 275, 282, 328, 331, 354, 366,389, 406, 409, 537, 538, 540, 542, 544),苏氨酸(Thr)含有8个位点(79, 132, 154, 228, 297, 302, 358, 440),络氨酸(Tyr)有6个位点(171, 220, 312, 333, 410, 434)。其中,537~544 aa为丝氨酸位点富集区。B细胞抗元表位分析结果:含有7个主要抗原表位(219~240, 300~314, 322~336, 352~372, 386~394, 500~508 和 533~547)。经PROSITE在线分析发现,在323~328、326~331和438~443存在3个可能的N端豆蔻酰基化位点(图3)。
|
图3 SRBSDV-HuNDC S10 氨基酸序列磷酸化位点预测
Figure 3 Predicted phosphorylation sites in amino acid sequence of SRBSDV-HuNDC S10
|
2讨论
SRBSDV是近年来发现的一种新病毒,目前的研究十分有限。因此,对各地该病毒衣壳蛋白编码基因的测定及其生物信息学分析很有必要,可为今后进一步阐释SRBSDV侵染细胞机制提供理论依据,为防治南方水稻黑条矮缩病提供有益借鉴。本研究成功测定了湖南鼎城病毒分离物SRBS-DV S10片段的完整序列,并对其进行了生物信息学分析。结果发现:SRBSDV-HuNDC S10两端存在保守序列和紧接保守序列的完全或不完全的反向互补序列,与前人报道的一致(Zhang et al., 2008; Wang et al., 2010)。更加特殊的是,这种结构不仅在该属其它病毒(除OSDV)衣壳蛋白序列上存在(表1) (McMa-hon et al., 1999),而且在SRBSDV的S1~S9片段上也均存在(Wang et al., 2010)。Kudo等(1991)曾推测,与SRBSDV同科的植物呼肠孤病毒属(Phytoreovirus)正链的3'端在负链RNA合成的起始过程中起了重要作用。考虑到该结构分布的特殊性和广泛性,笔者推测SRBSDV的末端序列也可能与负链RNA合成起始,进而与病毒的复制机制有关。有意思的是,在分析过程中发现唯独OSDV的保守区域与SRBSDV的保守区域“AAGTTTT”、“GCAGCTGATGTC”有很大不同,然而更有趣的是,若将SRBSDV S10两端互补区周围序列互换一段,将变为接近OSDV S8的两端序列(图4)。
|
图4 SRBSDV-HuNDC S10 两端序列变换后与OSDV S8 两端序列的比较
Figure 4 Comparison of the transformed SRBSDV-HuNDC S10 ends sequence and OSDV S8 ends sequence
|
这说明,两者保守区虽然不同,但还是具有一定的联系。由上分析可知,末端保守结构很可能与病毒复制有关,OSDV 是否为该属某种病毒复制过程出现差错而导致的产物,值得深究。
对SRBSDV S10的aa序列进行了糖基化位点、磷酸化位点及B细胞抗原表位的预测,对研究该蛋白的表达后蛋白修饰有重要的参考价值。据报道,正呼肠孤病毒(Orthoreovirus)外层衣壳μ1蛋白裂解释放出的N端豆蔻酰基化位点是该病毒穿过细胞膜屏障的必需部分(Liemann et al., 2002)。而SRBSDV衣壳蛋白发现的3个可能的N端豆蔻酰基化位点的生物学意义还不清楚,也可能与SRBSDV穿过细胞膜有关。本研究还对其编码的aa序列与部分Fijiviruses病毒对应片段aa序列进行了motif查找,分析了查找到的10 个保守区域在各病毒aa序列上的位置差异,为进一步研究蛋白质结构提供理论依据。
S10编码病毒的最外层衣壳蛋白,对其进行测定和生物信息学分析,丰富其分子水平资料,有助于对该病毒复制机制、侵染方式和致病性的深入研究,为防治SRBSDV提供理论依据。
3材料与方法
3.1植物材料
疑似感染SRBSDV水稻于2010年6月采自湖南省常德市鼎城区,保存于-20℃冰箱中。
3.2总RNA提取
所用的玻璃、瓷器均采用干热(180℃)灭菌4 h以上;配试剂等均用0.1% DEPC处理水。取50~100 mg病茎叶组织加入液氮充分研磨,迅速将其移入灭菌的1.5 mL离心管中。加入1 mL的Trizol试剂;加入500 mL氯仿-异戊醇(24:1)剧烈震荡摇匀15 s;冰浴10 min,12 000 r/min离心15 min,取上清;加入等体积的氯仿-异戊醇(24:1)轻微混匀,取上清;加入500 μL的异丙醇,颠倒混匀;室温放置10 min,12 000 r/min离心10 min,弃上清;加80%的乙醇洗涤沉淀,7 500 r/min离心5 min,弃上清;加无水乙醇洗涤沉淀,7 500 r/min离心5 min,弃上清;室温下沉淀并充分干燥后;溶于20 μL DEPC水中,-20℃保存备用。
3.3 SRBSDV的RT-PCR检测
用提取到的植株总RNA用特异性引物做RT-PCR(周倩等, 2010)。
RT反应体系:5 ×first stand Buffer 4 μL,dNTPs (10 mmol/L) 2 μL,F端引物(10 μmol/L) 1 μL,R端引物(10 μmol/L) 1 μL,M-MLV反转录酶(200 U/μL) 1 μL,总RNA 3 μL,DEPC处理水补足总体积20 μL。RT反应程序:65℃ 5 min,冰上孵育5 min;30℃ 10 min;42℃温浴20 min;99℃酶灭活5 min,立即至于冰上,4℃保存备用。
PCR反应体系:10×PCR Buffer 2.5 μL,F端引物(10 μmol/L) 1 μL,R端引物(10 μmol/L) 1 μL,dNTPs (10 mmol/L) 0.5 μL,Taq DNA聚合酶(2.5 U/μL) 0.5 μL,模板cDNA 2 μL,超纯水补足总体积25 μL。PCR扩增程序为:95℃ 5 min,95℃ 30 s,55℃ 45 s,72℃ 45 s, 35个循环后72℃延伸10 min。1.2%琼脂糖凝胶电泳检测扩增结果。
3.4双链RNA提取
参考Zhang等(2008)改良的硝酸纤维素CF-11亲和层析法从水稻病株叶组织中抽提病毒dsRNA(Dodds et al., 1984),经1%琼脂糖凝胶电泳检测后于-20℃保存备用。
3.5 SRBSDV S10全序列测序
为确保SRBSDV S10两端序列的准确性,用T4 RNA ligase(宝生物)在双链RNA的3'端加上接头引物zhm-1(表4) (Zhang et al., 2008)。
|
表4 扩增SRBSDV-HuNDC S10 的引物
Table 4 Primers used for amplifying SRBSDV-HuNDC S10 引物
|
RT反应体系:5 ×first stand Buffer 4 μL,dNTPs (10 mmol/L) 2 μL,zhm-2 (表4) (Zhang et al., 2008) (10 μmol/L) 2 μL,M-MLV反转录酶(200 U/μL) 1 μL,双链RNA 3 μL,超纯水补足总体积20 μL。RT反应程序:42℃温浴1 h。
PCR反应体系:2.5 μL 10 ×PCR Buffer,引物0.3 μmol/L (表4),dNTPs (2.5 mmol/L) 0.2 mmol/L,MgSO4 1.5 mmol/L,模板cDNA 0.5 μL,KOD DNA聚合酶 0.5 μL,超纯水补足总体积25 μL。PCR扩增程序为:98℃ 2 min,94℃ 10 s,55℃ 30 s,68℃ 1 min,35个循环后 68℃延伸10 min。
对PCR产物回收、连接、转化到pGM-T载体上,再经过蓝白斑筛选,挑取白色菌落,测定了两端 序列。同法,再用S10(1)-F、S10(1)-R对其中间片段 进行测序,得到SRBDV-HuNDC S10全序列。
3.6 S10同源性分析
应用DNASTAR Lasergene软件中的Megalign程序的Clustal W Method将湖南株S10与GenBank数据库上发布的9个相关毒株相应的核酸、氨基酸序列进行相似性比对。
同源性比对的相关毒株为:SRBSDV-r-GD株(EU784840);SRBSDV-r-HaN株(EU523360);SRBS-DV-m-YN株(HQ394212);RBSDV S10株(NC_0037-33);MRDV S10株(L76560);MRCV S10株(AY6075-86);OSDV S8株(AB011025);FDV S10 (NC_007162);NLRV S8株(NC_003653)。海南两个毒株S10全序列一致,仅选一个进行分析,山东玉米上SRBSDV分离物的S10全序列(GQ472845)NCBI数据库已收录,但 未公布,未作分析。
3.7 S10编码蛋白的motif分析
应用MEME软件(Bailey and Elkan, 1994) (http:// meme.sdsc.edu/meme/cgi-bin/meme.cgi)在线分析湖南株S10编码的氨基酸序列。参数设置为Distribution of motif occurrences: One per sequence; Number of di- fferent motifs: 10; Minimum motif width: 6; Maximum motif width: 50。
3.8序列比对和系统发育树的建立
应用Clustal X(version 1.83)的Alignment程序分别对10条序列进行多重对位排列(multiple alignments),使用MEGA4.1软件进行系统发育分析和遗传进化树的构建。遗传距离计算采用Kimura2-pa-rameter模式,并将对位排列中的缺失数据(missing data)或空位(gaps)完全删除(complete deletion),用邻位相连法(NJ, neighbor-joining)分析进化距离,并进 行自展(bootstrap)检验,重复1 000次。
3.9糖基化位点、磷酸化位点及B细胞抗原表位预测
Phos 2.0 (http://www.cbs.dtu.dk/services/NetPhos/)在线;使用PROSITE数据库(http://www.expasy.org/prosite)在线搜索可能的N端豆蔻酰基化位点。
作者贡献
该研究的实验和文章的写作部分由崔亚和朱俊子共同完成;高必达老师负责文章研究的组织、构思和技术指导;周倩老师负责论文修改工作,并对本实 验工作的顺利进行给予了大力支持和协调。
致谢
诚挚感谢湖南省常德市鼎城区植保站送检的疑似感染SRBSDV的水稻材料。
参考文献
Bailey T.L., and Elkan C., 1994, Fitting a mixture model by expectation maximization to discover motifs in biopolymers, Proc. Int. Conf. Intell. Syst. Mol. Biol., 2: 28-36
Dodds J.A., Morris T.J., and Jordan R.L., 1984, Plant viral double-stranded RNA, Annual Review of Phytopathology, 22: 151-168
Kudo H., Uyeda I., and Shikata E., 1991, Viruses in the Phytoreovirus genus of the Reoviridae family have the same con- served terminal sequences, Journal of General Virology, 72: 2857-2866
Liemann S., Chandran K., Baker T.S., Nibert M.L., and Harrison S.C., 2002, Structure of the reovirus membrane-penetration protein, Mu1, in a complex with is protector protein, Sig- ma3, Cell, 108(2): 283-295
McMahon J.A., and Dale J.L., and Harding R.M., 1999, Taxonomic implications for Fijiviruses based on the terminal sequences of Fiji disease fijivirus, Arch. Virol., 144 (11): 2259-2263
Milne R.G., del Vas M., Harding R.M., Marzachi R., and Mertens P.P.C., 2005, Genus fijivirus, In: Fauquet C.M., Mayo M.A., Maniloff J., Desselberger U., and Ball L.A. (eds.), Virus taxomony: Classification and nomenclature of viruses, Eighth Report of the International Committee on the Taxonomy of Viruses, Elsevier Academic Press, San Diego, pp.534-542
Wang Q., Yang J., Zhou G.H., Zhang H.M., Chen J.P., and Adams M.J., 2010, The complete genome sequence of two isolates of southern rice black-streaked dwarf virus, a new member of the genus Fijivirus, Journal of Phytopathology, 158(11-12): 733-737
Yin X., Xu F.F., Zheng F.Q., Li X.D., Liu B.S., and Zhang C.Q., 2011, Molecular characterization of segments S7 to S10 of a southern rice black-streaked dwarf virus isolate from maize in northern China, Virologica Sinica, 26(1): 47-53
Zhang H.M., Yang J., Chen J.P., and Adams M.J., 2008, A black-streaked dwarf disease on rice in China is caused by a novel Fijivirus, Archives of Virology, 153(10): 1893-1898
Zhou G.H., Wen J.J., Cai D.J., Li P., Xu D.L., and Zhang S.G., 2008, Southern rice black-streaked dwarf virus: A new pro- posed Fijivirus species in the family Reoviridae, Chinese Science Bulletin, 53(23): 3677-3685
Zhou G.H., Zhang S.G., Zou S.F., Xu Z.W., and Zhou Z.Q., 2010, Occurrence and damage analysis of a new rice dwarf disease caused by southern rice black-streaked dwarf virus, Zhiwu Baohu (Plant Protection), 36 (2): 144-146 (周国辉, 张曙光, 邹寿发, 许兆伟, 周志强, 2010, 水稻新病害南方水稻黑条矮缩病发生特点及危害趋势分析, 植物保护, 36(2): 144-146)
Zhou Q., Zhu J.Z., Liang J.G., Chen X.Y. and Gao B.D., 2010, Rapid detection of southern rice black-streaked dwarf virus, Jiyinzuxue Yu Yingyong Shengwuxue (Genomics and Ap- plied Biology), 29 (5): 1009-1012 (周倩, 朱俊子, 梁晋刚, 陈欣怡, 高必达, 2010, 南方水稻黑条矮缩病毒快速检测, 基因组学与应用生物学, 29(5): 1009-1012)
.png)
.png)
.png)

.png)
.png)
.png)
